| Pirrolisina | |
|---|---|
 | |
 | |
| Nome IUPAC | |
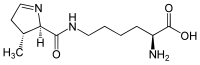
| N6-{[(2R,3R)-3-metil-3,4-diidro-2H-pirrol-2-il]carbonil}-L-lisina | |
| Abbreviazioni | |
| PYL, O | |
| Caratteristiche generali | |
| Formula bruta o molecolare | C12H21N3O3 |
| Massa molecolare (u) | 255,31 |
| Aspetto | ??? |
| Numero CAS | |
| PubChem | 5460671 e 23657811 |
| SMILES | CC1CC=NC1C(=O)NCCCCC(C(=O)O)N |
| Proprietà chimico-fisiche | |
| Costante di dissociazione acida a ??? K | pK1: ??? pK2: ??? |
| Punto isoelettrico | ??? |
| Solubilità in acqua | ??? |
| Temperatura di fusione | ??? |
| Indicazioni di sicurezza | |
| Frasi R | -- |
| Frasi S | -- |
La pirrolisina (PYL, O) è un amminoacido naturale, osservato per la prima volta nel 2004 in un archaebatterio, il Methanosarcina barkeri. Esso è il 22° amminoacido proteinogenico conosciuto, e la sua scoperta rimette in dubbio la marginalità del 21°, la selenocisteina. La pirrolisina è un derivato della lisina. Come la selenocisteina, è codificato da un codone di stop, il codone UAG, che in presenza di particolari segmenti di RNA viene interpretato come elemento costitutivo.
Incorporazione in proteine
[modifica | modifica wikitesto]La pirrolisina è sintetizzata in vivo tramite la condensazione di una molecola di L-lisina e una di 3-metilornitina [1]
L'incorporazione della pirrolisina all'interno di proteine non avviene attraverso dei meccanismi di modificazione post-traduzionale, ma tramite il classico processo di sintesi proteica. In Methanosarcina sp. il riconoscimento del codone AUG è mediato dai geni pylT e pylS i quali codificano rispettivamente un particolare tRNA dotato dell'anticodone CUA e un amminoacil-tRNA sintetasi dedicato a caricare la pirrolisina sul corrispondente tRNA [1].
La scoperta che il sistema di incorporazione della pirrolisina non influisce sui processi metabolici endogeni di Escherichia coli, in gergo pertanto definito ortogonale, ha permesso il suo utilizzo per incorporare amminoacidi non canonici all'interno di proteine[2].
Ruolo biologico
[modifica | modifica wikitesto]L'anello pirrolico aggiuntivo presente sulla catena laterale della pirrolisina sembra abbia un ruolo catalitico all'interno del sito attivo di numerose metiltransferasi [3]. Esso è in grado di posizionare precisamente il gruppo metile di una metilammina in maniera tale da consentire l'attacco di un cofattore corrinoide. Il modello proposto suggerisce che un residuo amminoacidico in possesso di un gruppo carbossilico, ad esempio il glutammato, trasferisca un protone all'azoto presente sull'anello pirrolico. Ciò permette alla metilammina di effettuare una addizione nucleofila sull'atomo di carbonio adiacente all'azoto protonato sull'anello. La diversa carica presente sull'atomo di azoto (positivo) e sul gruppo carbossilico del glutammato (negativo) spinge l'addotto anello pirrolico/metilammina a riorientarsi nel sito attivo portando così il gruppo metilico ad entrare in contatto col cofattore corrinoide. Ciò permette uno scambio netto di CH+3 dalla metilammina al metallo presente nel cofattore, ad esempio cobalto, il quale passa da uno stato di ossidazione I a III. Questo processo produce ammoniaca come prodotto di scarto, la quale viene poi rilasciata per poter riciclare l'enzima in attesa della prossima reazione [3].
Note
[modifica | modifica wikitesto]- ^ a b Marsha A. Gaston, Liwen Zhang, Kari B. Green-Church e Joseph A. Krzycki, The complete biosynthesis of the genetically encoded amino acid pyrrolysine from lysine, in Nature, vol. 471, 30 marzo 2011, pp. 647–650, DOI:10.1038/nature09918.
- ^ Ana Crnković, Tateki Suzuki, Dieter Söll e Noah M. Reynolds, Pyrrolysyl-tRNA synthetase, an aminoacyl-tRNA synthetase for genetic code expansion, in Croatica Chemica Acta, vol. 89, n. 2, giugno 2016, pp. 163–174, DOI:10.5562/cca2825.
- ^ a b Bing Hao, Gong, Ferguson, James, Krzycki e Chan, A New UAG-Encoded Residue in the Structure of a Methanogen Methyltransferase, in Science, vol. 296, n. 5572, 24 maggio 2002, pp. 1462–1466, DOI:10.1126/science.1069556, PMID 12029132.
Bibliografia
[modifica | modifica wikitesto]- John F. Atkins and Ray Gesteland, The 22nd Amino Acid, in Science, vol. 296, n. 5572, 2002, pp. 1409–1410, DOI:10.1126/science.1073339, PMID 12029118.
- Krzycki J, The direct genetic encoding of pyrrolysine, in Curr Opin Microbiol, vol. 8, n. 6, 2005, pp. 706–712, DOI:10.1016/j.mib.2005.10.009, PMID 16256420.
Altri progetti
[modifica | modifica wikitesto] Wikimedia Commons contiene immagini o altri file su Pirrolisina
Wikimedia Commons contiene immagini o altri file su Pirrolisina








