RNA enhancer
Gli RNA enhancer (eRNA) rappresentano una classe di molecole di RNA non codificanti relativamente lunghe (50-2000 nucleotidi) trascritte dalla sequenza di DNA delle regioni enhancer (intensificatrici). Sono stati rilevati per la prima volta nel 2010, attraverso l'uso di tecniche quali RNA-seq e ChIP-seq[1] . Gli eRNA possono essere suddivisi in due classi principali: eRNA 1D ed eRNA 2D, che differiscono principalmente in termini di dimensioni, stato di poliadenilazione e direzionalità trascrizionale.[2] L'espressione di un dato eRNA è correlata con l'attività del suo corrispondente enhancer nei geni bersaglio.[3] Un numero sempre maggiore di studii suggerisce che gli eRNA svolgono un ruolo attivo nella regolazione trascrizionale in cis e in trans e, sebbene i loro meccanismi d'azione rimangano poco chiari, sono stati proposti alcuni modelli.[2]
Scoperta
[modifica | modifica wikitesto]gli enhancer come siti di trascrizione extragenica sono stati inizialmente scoperti in studi sull'intero genoma, che hanno identificato gli enhancer come regioni comunemente presentanti una capacità di legame dell'RNA polimerasi II (RNA pol II) e di trascrizione di RNA non codificanti per proteine.[4][1] Il livello di interazione RNA pol II-enhancer e la formazione di trascritti di RNA sono risultati altamente variabili tra questi studi iniziali. Utilizzando picchi espliciti della firma della cromatina, è stata trovata una proporzione significativa (~ 70%) di siti di inizio della trascrizione di RNA Pol II extragenici sovrapposti a siti di potenziamento nei macrofagi murini .[4] Su 12.000 potenziatori neuronali nel genoma del topo, è stato scoperto che quasi il 25% dei siti lega l' RNA Pol II e genera trascrizioni .[5] Questi eRNA, a differenza degli RNA messaggeri (mRNA), non erano modificati mediante poliadenilazione, erano generalmente brevi e non codificanti e venivano trascritti in modo bidirezionale. Studi successivi hanno rivelato la trascrizione di un altro tipo di eRNA, generato tramite trascrizione unidirezionale, che era più lungo e conteneva una coda di poliA.[6] Inoltre, i livelli di eRNA sono stati correlati con i livelli di mRNA dei geni vicini, suggerendo il potenziale ruolo regolatorio e funzionale di queste molecole di RNA potenziatore non codificante.[1]
biogenesi
[modifica | modifica wikitesto]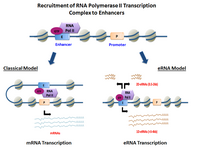
Riepilogo
[modifica | modifica wikitesto]Gli eRNA sono trascritti da sequenze di DNA a monte e a valle di regioni enhancer extrageniche.[7] In precedenza, diversi enhancer modello hanno dimostrato capacità di reclutare direttamente RNA Pol II e fattori di trascrizione generali per formare il complesso di pre-inizio (PIC) a livello del sito di inizio sul promotore dei vari geni . In alcuni tipi di cellule , gli enhancer attivati hanno dimostrato la capacità sia di reclutare RNA Pol II sia di fornire uno stampo per la trascrizione attiva delle loro sequenze locali.[4][1]
A seconda della direzionalità della trascrizione, le regioni enhancer generano due diversi tipi di trascritti non codificanti, 1D-eRNA e 2D-eRNA. La natura delle pre-iniziazione complessi e specifici fattori di trascrizione reclutati al enhancer può controllare il tipo di eRNA generato. Dopo la trascrizione, la maggior parte degli eRNA rimane nel nucleo .[8] In generale, gli eRNA sono molto instabili e attivamente degradati dall'esosoma nucleare. Non tutti i potenziatori sono trascritti, con potenziatori non trascritti che superano di gran lunga quelli trascritti nell'ordine di grandezza di dozzine di migliaia in ogni dato tipo di cellula.[4]
eRNA 1D
[modifica | modifica wikitesto]Nella maggior parte dei casi, la trascrizione unidirezionale delle regioni enhancer genera eRNA lunghi (>4kb) e poliadenilati. Gli enhancer che generano eRNA poliA+ hanno un rapporto H3K4me1 /me3 più basso nella loro firma cromatinica rispetto agli eRNA 2D.[6] Gli eRNA PolyA+ sono distinti dai lunghi poli-trascritti multiesonici (meRNA), che sono generati dalla trascrizione di enhancer intragenici. Questi lunghi RNA non codificanti, che riflettono accuratamente la struttura del gene ospite ad eccezione del primo esone alternativo, mostrano uno scarso potenziale di codifica.[9]
eRNA 2D
[modifica | modifica wikitesto]La trascrizione bidirezionale nelle regioni enhancer genera eRNA relativamente più corti (0,5-2 kb) e non poliadenilati. Gli enhancer che generano poliA-eRNA hanno una firma della cromatina con un rapporto H3K4me1/me3 più elevato rispetto agli 1D-eRNA. In generale, la trascrizione degli enhancer e la produzione di eRNA bidirezionali dimostrano una forte correlazione dell'attività dell'enhancer sulla trascrizione genica .[10]
Meccanismi di funzionamento proposti
[modifica | modifica wikitesto]
L'idea che non tutti gli enhancer vengano trascritti contemporaneamente e che la trascrizione degli eRNA sia correlata all'attività specifica dell'enhancer supporta l'idea che i singoli eRNA svolgano funzioni biologiche distinte e rilevanti.[2] Tuttavia, non c'è ancora consenso sul significato funzionale degli eRNA. Inoltre, gli eRNA possono essere facilmente degradati attraverso esosomi e decadimento mediato da un nonsenso (NMD), il che limita il loro potenziale come importanti regolatori trascrizionali.[11] Ad oggi sono stati proposti quattro principali modelli di funzione dell'eRNA,[2] ciascuno supportato da diverse linee di evidenza sperimentale.
Rumore trascrizionale
[modifica | modifica wikitesto]Poiché numerosi studi hanno dimostrato che l' RNA Pol II può trovarsi spesso in un numero molto elevato di regioni extrageniche, è possibile che gli eRNA rappresentino semplicemente il prodotto di una "gocciolante" trascrizione casuale e non abbiano alcun effettivo significato funzionale.[4] L'attività non specifica di RNA Pol II consentirebbe quindi rumore trascrizionale extragenico nei siti in cui la cromatina è già in uno stato aperto e trascrizionalmente competente. Ciò spiegherebbe anche l'espressione di eRNA tessuto-specifica[12] in quanto anche i siti aperti sono tessuto-specifici.
Effetti dipendenti dalla trascrizione
[modifica | modifica wikitesto]La trascrizione genica mediata da RNA Pol II induce un'apertura locale dello stato della cromatina attraverso il reclutamento di istone acetiltransferasi e altri modificatori dell'istone che promuovano la formazione di eucromatina. È stato proposto che la presenza di questi enzimi potrebbe anche indurre un'apertura della cromatina nelle regioni enhancer, che di solito sono presenti in località distanti ma possono essere reclutate per attivare la trascrizione dei vari geni attraverso il looping del DNA .[13] In questo modello, gli eRNA sono quindi espressi in risposta alla trascrizione di RNA Pol II e dunque non svolgono alcuna funzione biologica.
Attività funzionale in cis
[modifica | modifica wikitesto]Mentre i due modelli precedenti implicavano che gli eRNA non fossero funzionalmente rilevanti, questo meccanismo afferma che gli eRNA sono molecole funzionali che mostrano attività cis. In questo modello, gli eRNA possono reclutare localmente proteine regolatrici nel proprio sito di sintesi. A sostegno di questa ipotesi, si pensa che i trascritti degli enhancer a monte del gene della ciclina D1 (G1) servano come adattatori per il reclutamento delle istone acetiltransferasi. È stato infatti scoperto che la deplezione di questi eRNA porta al silenziamento trascrizionale della ciclina D1.[8]
Attività funzionale in trans
[modifica | modifica wikitesto]L'ultimo modello prevede la regolazione trascrizionale da parte degli eRNA in posizioni cromosomiche distanti. Attraverso il reclutamento differenziale di complessi proteici, gli eRNA possono influenzare la competenza trascrizionale di loci specifici. Evf-2 rappresenta un buon esempio di eRNA con azione regolatoria in trans, in quanto può indurre l'espressione di DLX2, che a sua volta può aumentare l'attività degli enhancer DLX5 e DLX6.[14] Gli eRNA agenti in trans potrebbero anche funzionare in cis e viceversa.
Rilevamento sperimentale
[modifica | modifica wikitesto]Il rilevamento degli eRNA è abbastanza recente (2010) ed è stato reso possibile tramite indagini sull'intero genoma, come il sequenziamento dell'RNA (RNA-seq) e il sequenziamento dell'immunoprecipitazione della cromatina (ChIP-seq).[1] RNA-seq consente l'identificazione diretta degli eRNA abbinando il trascritto rilevato alla corrispondente sequenza di enhancer attraverso analisi bioinformatiche.[15] ChIP-seq rappresenta un modo meno diretto per valutare la trascrizione di un enhnancer, ma può anche fornire informazioni cruciali rilevando le specifiche firme cromatiniche che sono associate agli enhancer attivi.[16] Sebbene alcuni dati rimangano controversi, il consenso in letteratura è che la migliore combinazione di modificazioni post-traduzionali degli istoni sugli enhancerattivi sia costituita da H2AZ, H3K27ac e un alto rapporto di H3K4me1 su H3K4me3.[16][17][18] Gli esperimenti con ChIP possono essere condotti anche con anticorpi che riconoscano l' RNA Pol II, che si trova nei siti in cui la trascrizione è attiva.[4] La rilevazione sperimentale degli eRNA è complicata dalla loro bassa stabilità endogena a cause della degradazione mediata da esosoma e dal decadimento mediato da un nonosenso (NMD) .[11] Il saggio "run-on", che utilizza una tecnica di sequenziamento ad alto rendimento, è un altro modo per rilevare gli eRNA. Il sequenziamento Precision Run-On (PRO-seq) è in grado di rilevare gli RNA che vengono trascritti attivamente e ha una risoluzione fino a una coppia di basi.[19] Il fatto che gli eRNA tendano ad essere espressi da enhancer attivi potrebbe rendere la loro rilevazione uno strumento utile per distinguere tra potenziatori attivi e inattivi.
Implicazioni nello sviluppo e nella patologia
[modifica | modifica wikitesto]La prova che gli eRNA causino effetti a valle sull'efficienza dell'attivazione dell'enhancer e sulla trascrizione genica suggerisce le loro capacità funzionali e la loro potenziale importanza. È stato dimostrato che il fattore di trascrizione p53 possa legare le regioni enhancer e generare eRNA in modo p53- dipendente.[20] Nel cancro, p53 svolge un ruolo centrale nella soppressione del tumore ed è stato dimostrato che le mutazioni del gene compaiono nel 50% dei tumori .[21] È stato dimostrato anche che queste regioni ehnancer legate a p53 (p53BER) interagiscono poi con più bersagli genici locali e distali, coinvolti nella proliferazione cellulare e sopravvivenza cellulare. Inoltre, si è visto che gli eRNA generati dall'attivazione di p53BER paiono essere necessari per una trascrizione efficiente dei geni bersaglio di p53, indicando il loro importante probabile ruolo regolatore degli eRNA nella soppressione del tumorale e nel cancro.
Variazioni negli enhancer sono state collegate ad alcune patolgie umane, ma attualmente non sembra possibile un approccio terapeutico per manipolare l'attivtà degli enhancer. Con l'emergere di eRNA come componenti importanti nell'attività dell'enhancer, strumenti terapeutici potenti come l'RNAi potrebbero fornire percorsi promettenti per colpire l'interruzione dell'espressione genica.
Note
[modifica | modifica wikitesto]- ^ a b c d e Widespread transcription at neuronal activity-regulated enhancers, in Nature, vol. 465, n. 7295, May 2010, pp. 182–7, DOI:10.1038/nature09033, PMID 20393465.
- ^ a b c d Noncoding transcription at enhancers: general principles and functional models, in Annual Review of Genetics, vol. 46, 2012, pp. 1–19, DOI:10.1146/annurev-genet-110711-155459, PMID 22905871.
- ^ Chromosome contacts in activated T cells identify autoimmune disease candidate genes, in Genome Biology, vol. 18, n. 1, September 2017, pp. 165, DOI:10.1186/s13059-017-1285-0, PMID 28870212.
- ^ a b c d e f A large fraction of extragenic RNA pol II transcription sites overlap enhancers, in PLoS Biology, vol. 8, n. 5, May 2010, pp. e1000384, DOI:10.1371/journal.pbio.1000384, PMID 20485488.
- ^ Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome, in Nature Genetics, vol. 39, n. 3, March 2007, pp. 311–8, DOI:10.1038/ng1966, PMID 17277777.
- ^ a b Transcription initiation platforms and GTF recruitment at tissue-specific enhancers and promoters, in Nature Structural & Molecular Biology, vol. 18, n. 8, July 2011, pp. 956–63, DOI:10.1038/nsmb.2085, PMID 21765417.
- ^ Molecular analysis of enhancer RNAs and chromatin modifications in the region of their synthesis in Drosophila cells possessing genetic constructs, in Doklady. Biochemistry and Biophysics, vol. 442, 2012, pp. 7–11, DOI:10.1134/S1607672912010012, PMID 22419084.
- ^ a b Induced ncRNAs allosterically modify RNA-binding proteins in cis to inhibit transcription, in Nature, vol. 454, n. 7200, July 2008, pp. 126–30, DOI:10.1038/nature06992, PMID 18509338.
- ^ Intragenic enhancers act as alternative promoters, in Molecular Cell, vol. 45, n. 4, February 2012, pp. 447–58, DOI:10.1016/j.molcel.2011.12.021, PMID 22264824.
- ^ Reprogramming transcription by distinct classes of enhancers functionally defined by eRNA, in Nature, vol. 474, n. 7351, May 2011, pp. 390–4, DOI:10.1038/nature10006, PMID 21572438.
- ^ a b Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase, in Cell, vol. 121, n. 5, June 2005, pp. 725–37, DOI:10.1016/j.cell.2005.04.030, PMID 15935759.
- ^ Transcription: Enhancers make non-coding RNA, in Nature, vol. 465, n. 7295, May 2010, pp. 173–4, DOI:10.1038/465173a, PMID 20463730.
- ^ The histone acetyltransferase PCAF associates with actin and hnRNP U for RNA polymerase II transcription, in Molecular and Cellular Biology, vol. 28, n. 20, October 2008, pp. 6342–57, DOI:10.1128/MCB.00766-08, PMID 18710935.
- ^ The Evf-2 noncoding RNA is transcribed from the Dlx-5/6 ultraconserved region and functions as a Dlx-2 transcriptional coactivator, in Genes & Development, vol. 20, n. 11, June 2006, pp. 1470–84, DOI:10.1101/gad.1416106, PMID 16705037.
- ^ RNA-Seq: a revolutionary tool for transcriptomics, in Nature Reviews. Genetics, vol. 10, n. 1, January 2009, pp. 57–63, DOI:10.1038/nrg2484, PMID 19015660.
- ^ a b High-resolution profiling of histone methylations in the human genome, in Cell, vol. 129, n. 4, May 2007, pp. 823–37, DOI:10.1016/j.cell.2007.05.009, PMID 17512414.
- ^ Discovery of active enhancers through bidirectional expression of short transcripts, in Genome Biology, vol. 12, n. 11, November 2011, pp. R113, DOI:10.1186/gb-2011-12-11-r113, PMID 22082242.
- ^ Histone H3K27ac separates active from poised enhancers and predicts developmental state, in Proceedings of the National Academy of Sciences of the United States of America, vol. 107, n. 50, December 2010, pp. 21931–6, DOI:10.1073/pnas.1016071107, PMID 21106759.
- ^ Base-pair-resolution genome-wide mapping of active RNA polymerases using precision nuclear run-on (PRO-seq), in Nature Protocols, vol. 11, n. 8, August 2016, pp. 1455–76, DOI:10.1038/nprot.2016.086, PMID 27442863.
- ^ eRNAs are required for p53-dependent enhancer activity and gene transcription, in Molecular Cell, vol. 49, n. 3, February 2013, pp. 524–35, DOI:10.1016/j.molcel.2012.11.021, PMID 23273978.
- ^ Live or let die: the cell's response to p53 (PDF), in Nature Reviews. Cancer, vol. 2, n. 8, August 2002, pp. 594–604, DOI:10.1038/nrc864, PMID 12154352.
Collegamenti esterni
[modifica | modifica wikitesto]- Vista Enhancer Database
- Mouse ENCODE Project Archiviato il 13 dicembre 2012 in Internet Archive.
- Progetto ENCODE all'UCSC
- PEDB








